本文分为两部分:SnapGene软件概览;如何使用SnapGene将DNA序列转换为氨基酸序列;
SnapGene软件概览
1.打开SnapGene软件,可以看到欢迎界面;
2.选择一个选项就可以进入到软件的界面;
3.这里,我们先打开一个已有的文件继续操作;
4.进入软件界面后,我们可以看到顶部的菜单和工具栏;
5.以及侧面的工具栏;
6.还有底部的选项卡菜单,可以预览不同的形式;
7.侧面的这两个工具栏提供了对常见文件操作和显示的快速访问:
8.底部的选项卡在注释中显示序列的备用视图;
9.和其他软件一样,SnapGene顶部的【File】“文件”菜单有创建、打开和保存文件的选项;
10.SnapGene顶部的工具栏里也包含了常用的功能;
11.在顶部的【Edit】“编辑”菜单中,包括了针对核酸(nucleic acids)和蛋白质(proteins)进行优化的常用编辑工具:
12.【View】“视图”菜单里有全部的查看和交互选项,允许用户在序列的不同视图之间切换、隐藏或显示工具栏。也可以从这里更改字体大小,或者打开和关闭注释。
注:SnapGene软件概览部分图片来源于网络
如何使用SnapGene将DNA序列转换为氨基酸序列
在序列比对的过程中,常常需要将核苷酸序列转换为氨基酸序列,比对氨基酸是否有所突变。方法有很多,我常用的一种是通过SnapGene软件转换。操作如下:
1.需要准备一条目的基因序列(fasta格式),通过SnapGene软件打开。点击Map选项卡,选中CDS的区域(如果这个文件为cds序列,则全部选中;若cds是片段,则选中cds的片段部分)
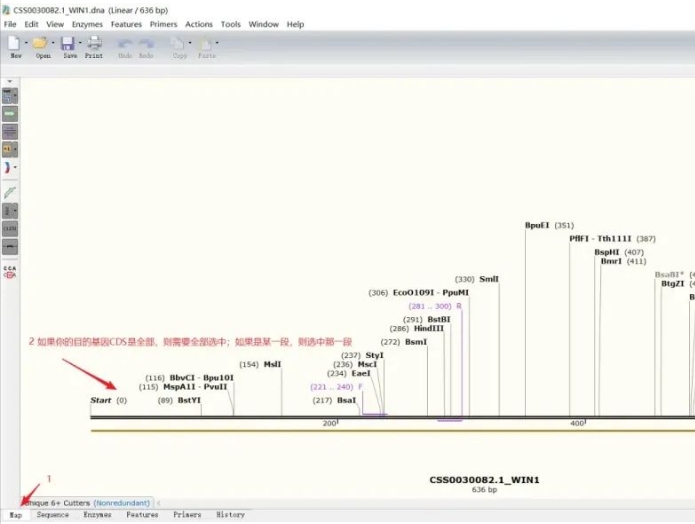
进入页面
2.点击上方feature-Add feature,给CDS区域加上feature;
3.设置feature,点击Type-CDS-OK,设置完毕;
4.点击Action-Make protein,得到氨基酸序列;
5.点击Sequence即是需要氨基酸的序列,可以保存为prot文件,也可以复制保存为fasta格式文件。
上一条:SnapGene 6.02科研软件主要功能
下一条:怎样将 MongoDB 转变为预测数据库?