做质粒图谱、直观的查找酶切位点、标记基因片段属性、直观查看引物所在位置,查看开放阅读框所编码氨基酸、直观的序列比对…… Snapgene简直就是做分子克隆的利器。
今天小编就以实验室用的经典版本为例,整理介绍一下snapgene的主要功能和操作流程,希望能帮到各位初接触分子克隆的同僚们。
01建立质粒分析图谱
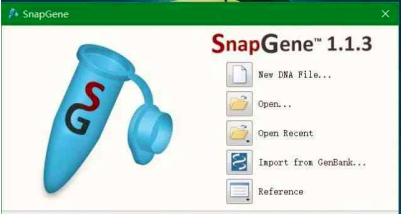
运行软件,界面如图所示。选择新建dna文件,将从网上得到的序列复制粘贴入框中,命好名字。对于质粒,Topology选择circular(环形),勾选Detect common features,将一些质粒共有的常见的特征加入其中,结果会如下图,
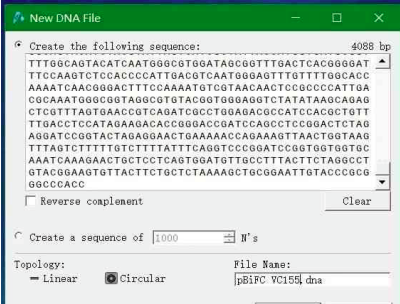
选择add 10 features 就可以生成一个高大上的质粒图谱了。当然,我们也可以直接在诸如addgene这样的网站内上下载现成的dna文件或者gbk文件啦~
(1)MAP视图
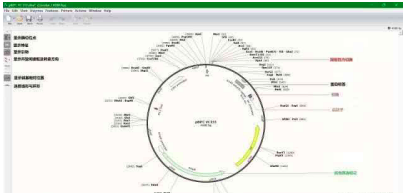
在这个视图上我们可以直观的看到很多的信息,包括酶切位点,启动子(promoter),增强子(enhancer),复制起始位点(ori),抗性筛选标记,HA等蛋白标签等等,点左侧的按钮可以选择显示不显示这些信息。在这个图上,酶切位点和引物的位置和相对关系等信息可以说是一目了然,这对于我们在建立克隆时选取酶切位点,测序鉴定克隆时选取测序引物都是十分便利的。
(2)sequence视图
图谱是在MAP视图下看到的结果,我们还可以在sequence视图下来分析序列。这个视图下我们可以很清楚看到序列信息,包括碱基序列信息、由不同的开放阅读框得到的氨基酸序列信息。在这里我们可以自由调整氨基酸的三字母缩写和单字母缩写形式。在这个视图下酶切位点、引物、蛋白标签等参考信息还都是存在的,因此我们在这个视图下设计引物会十分方便。
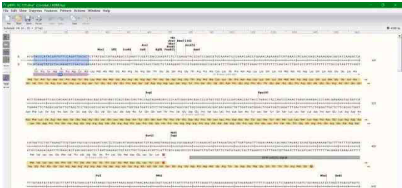
(3)Enzyme视图与feature视图
Enzyme视图下,我们可以看到各个内切酶的位点和数目,但小编认为这样反倒不如在图谱中看得更直接一些了。
Feature视图下,可以查看常见的的质粒共同特征,除了常见的这些,也可以自己人为的添加特征标签,可以自主的设计特征的类型,特征序列的大小,读取方向,起始位置是否分片段等等。
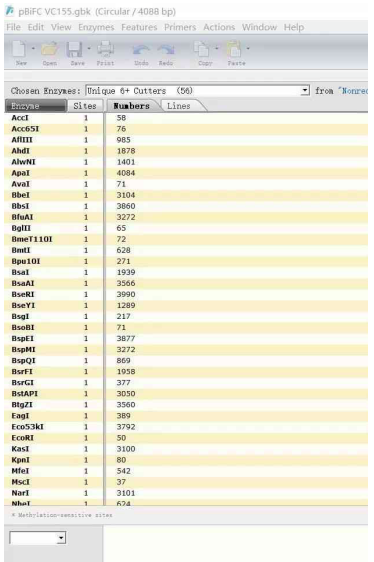
02设计PCR引物
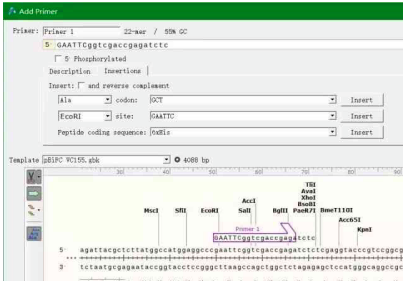
在多克隆位点处找到合适的两个酶切位点。在目的基因上下游截取15-30bp序列,点击Primers→Add primers。如图:
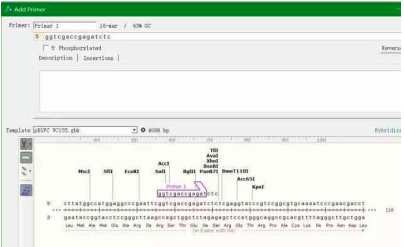
给该引物命名,然后点击Insertion,在该引物上添加之前选择好的酶切位点序列,如图选择了EcoR I,点击Insert即可完成(必要时需要加保护碱基),还可以在引物上添加多肽标签,如6×His等。最后点击add primer to template,即可完成引物设计与添加。
03序列比对
Snapgene具有序列比对的功能。当测序结果送到,我们需要比对检测我们建立的克隆是否序列正确。如下图所示,先在snapgene中打开你的测序结果,选择View → Align With A Sequence Trace, 然后选取你比对标准文件,比如你的目的基因序列,即可得到序列比对图。
如图是小编的一个测序结果,小编在某蛋白的基因上构建了两个氨基酸的点突变,对点突变质粒的测序比对结果发现有5个错配(mismatch)和1个间断(gap),这些会用红色标出。5个错配就是小编设计的突变位点,而除了这五个之外,其余都是一一对应,表明我点突变得到的质粒只在目标位置有突变,其余都正确,而1个间断是因为小编在蛋白的C端添加了flag标签,而目的蛋白基因到C端即直接是TGA终止密码子,所以有间断,这也是预期内的结果。所以小编建立的点突变是可用的啦~
04模拟建立克隆
Snapgene具有模拟建立克隆的功能,这使得我们设计建立克隆方案更加简便,如果克隆过程设计方案有缺陷,我们可以借助模拟发现并做出纠正。Snapgene可以模拟的标准限制性克隆也可以模拟融合克隆。步骤分别如下
(1)模拟标准限制性克隆:
①打开需要被插入片段的质粒图谱,点击Actions → insert fragments。
②切载体。在质粒中选取合适的酶切位点,单酶切直接点击该酶即可,双酶切则需要按住shfit按键再点击两个酶,这样即完成载体的酶切;
③切目的基因片段。选取insert→source of insert,将需要插入的目的基因片段序列文件加入其中,在目的基因片段中选取同样的两个酶进行酶切,必要时点击调整方向使其匹配,点击clone完成模拟。
(2)模拟融合克隆
打开需要被插入片段的质粒图谱,点击Actions →in-fusion ®cloning →insert one fragments。
①在vector 选项卡,选取要插入的位点或者要替换的区域。在这里,小编准备随意替换掉质粒上的抗性bla(β-内酰胺酶)区域,选取该段。
②在fragment选项卡,选替换片段,注意该片段阅读方向与被替换质粒位点的方向是否一致,如不一致,点击更换方向。这里小编选取卡那抗性。
③在product选项卡点击Choose Overlapping PCR Primers,即可设计好引物,形成融合后的质粒图谱。可以看到原来的β-内酰胺酶区域已经被替换成了卡那抗性标记。
若想获得引物的序列,点击Primers→Export Selected Primers,选择保存。
上一条:SnapGene业界受欢迎的分子克隆工具
下一条:Minitab将于2023年1月起执行新的价格